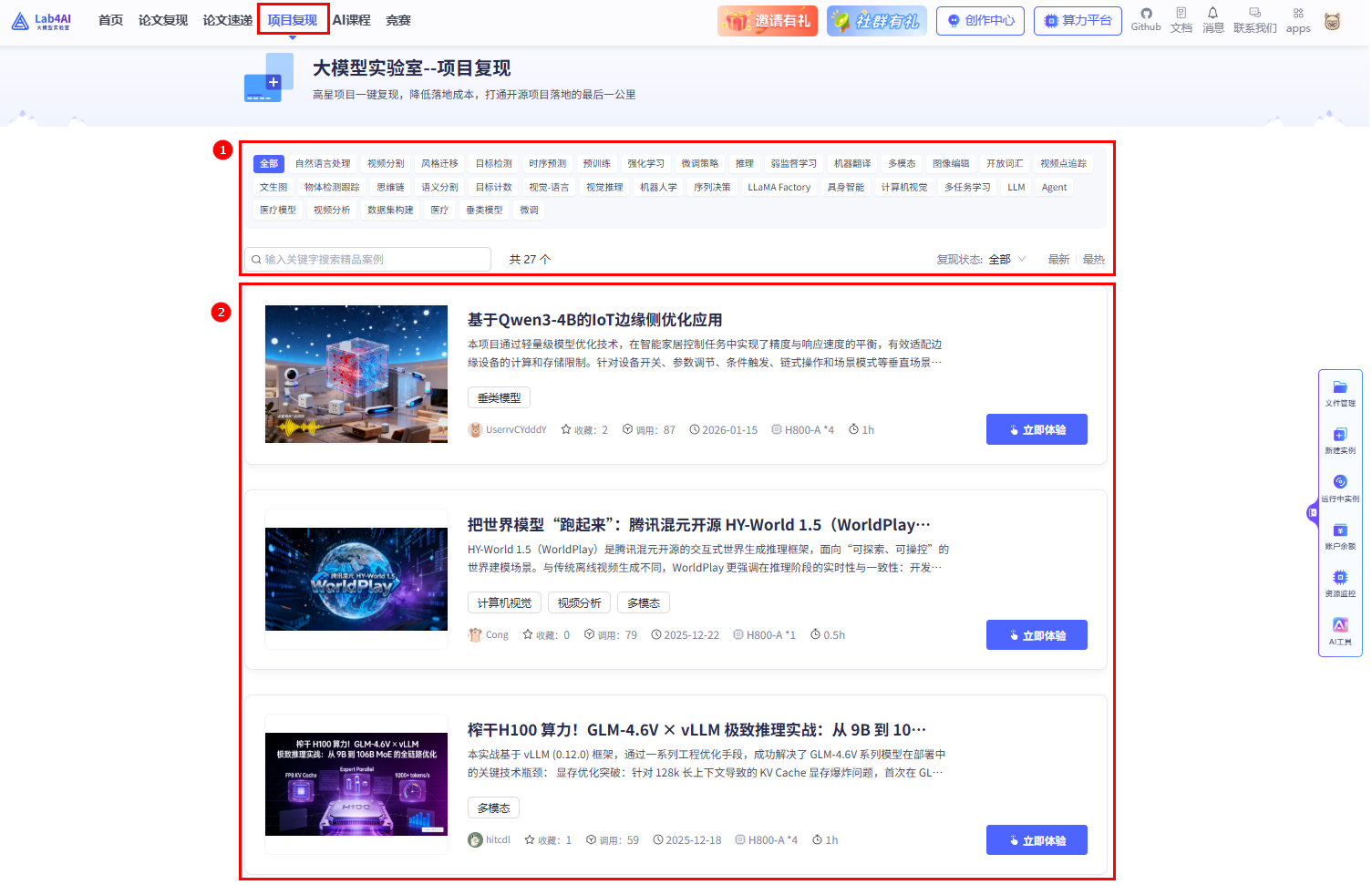
项目复现体验
大模型实验室提供“项目复现”功能模块,支持用户便捷地复现来自GitHub等开源社区的高星项目。平台集成高性能弹性算力资源,帮助用户快速构建开发环境,提升科研与开发效率。
前提条件
概览
项目复现界面主要由筛选区和项目列表两部分组成。
| 序号 | 模块名称 | 说明 |
|---|---|---|
| ① | 项目筛选 | 您可通过设置所属领域、关键词、复现状态等条件,对项目进行筛选。 |
| ② | 项目列表 | 您可以查看项目详情、原文内容以及 GitHub 代码仓库链接,也可点击“立即体验”按钮,快速复现项目。 |
操作步骤
-
登录大模型实验室,系统默认进入平台首页,点击[项目复现]菜单,跳转至项目复现列表页面,例如上图所示。以下操作以复现“DISC-MedLLM”项目为例进行说明。
-
找到“DISC-MedLLM”项目,点击“立即体验”按钮,选择JupyterLab、VSCode或LLaMA Factory Online实例。以JupyterLab为例,点击“JupyterLab”图标,进入资源配置页面,根据需求选择合适的资源规格和显卡数量,确认后点击“启动”,即可进入 JupyterLab 环境。
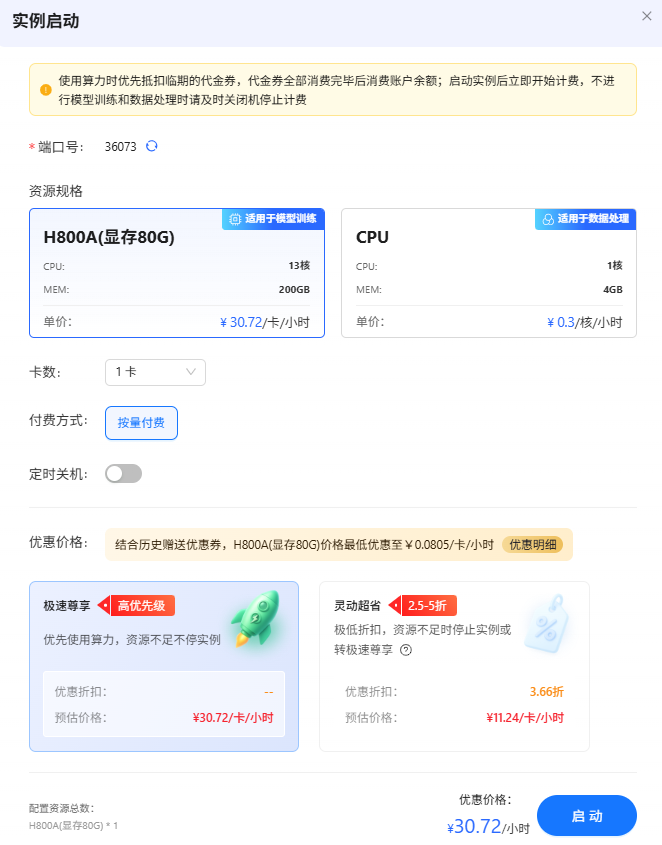 提示
提示LLaMA Factory Online类型的实例需要在管理后台配置,配置之后您可启动LLaMA Factory的WebUI实例,您可在实例内进行对应的模型训练、评估、对话、或者导出等操作。
-
进入
codelab/DISC-MedLLM/code目录,点击“Terminal”进入终端。您可选择使用系统预置的默认环境或自定义环境进行论文复现。该项目下的文件目录包括:code、dataset、model,具体说明如下表所示。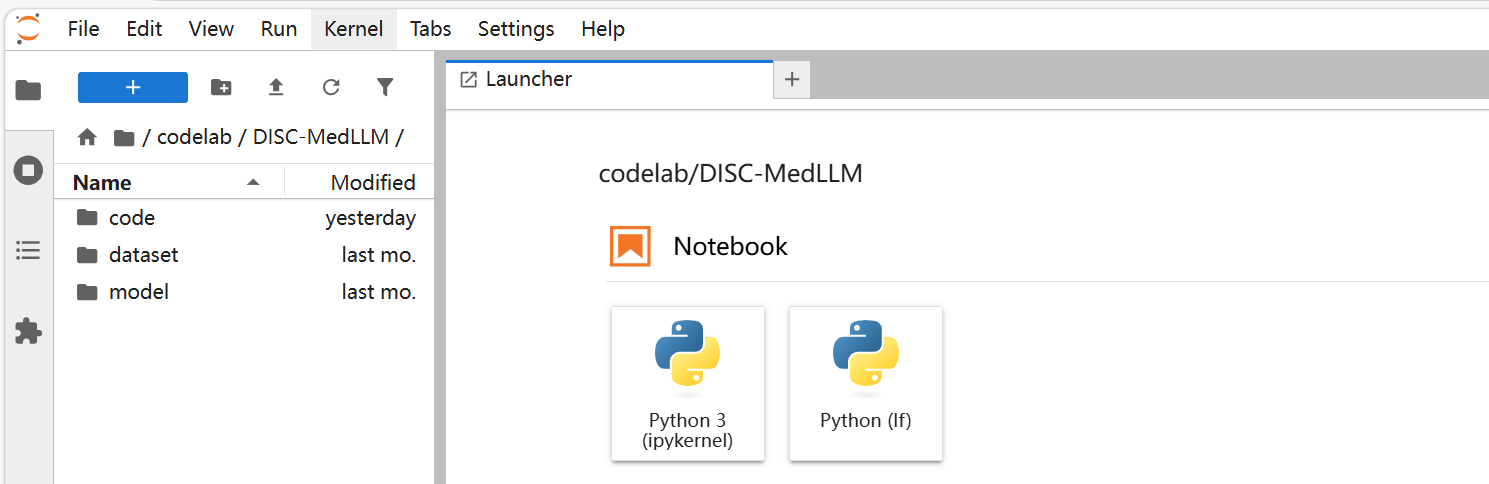
文件名称 说明 code 存储代码文件,可读取,可写入。 dataset 存储项目数据集,只可读。 model 存储项目模型,只可读。
- 默认环境
- 自定义环境
-
如果您使用默认环境进行项目复现,且为首次操作,请运行以下命令配置环境。
- 运行如下所示的命令激活系统内置的项目环境。
conda activate doodle- 运行如下所示的命令,在已经激活的Python环境中安装
ipykernel包。
pip install ipykernel -i https://pypi.tuna.tsinghua.edu.cn/simple- 运行如下所示的命令,将当前Python环境注册为Jupyter内核。
kernel_install --name doodle --display-name "python(doodle)"提示您需在对应的Conda环境中运行上述命令,否则内核(kernel)将无法正确注册。
-
刷新页面,单击
 图标返回Launcher页面,单击选择Notebook板块下已注册的环境,本例选择上步已激活的 "python(doodle)" 内核,例如下图所示。
图标返回Launcher页面,单击选择Notebook板块下已注册的环境,本例选择上步已激活的 "python(doodle)" 内核,例如下图所示。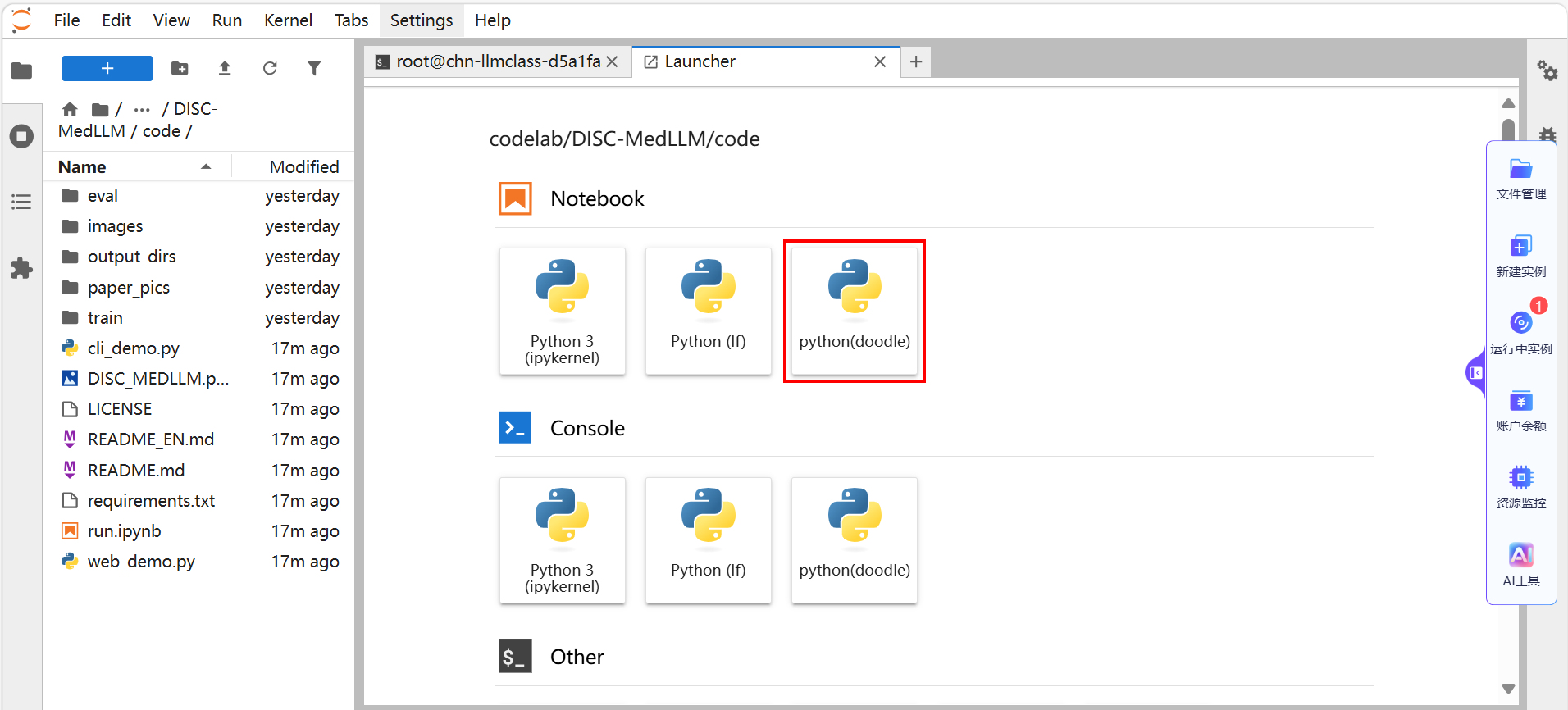
-
切换至
run.ipynb,如下图高亮①所示;点击右上角(如下图高亮②所示),选择“python(doodle)”环境,如下图高亮③所示;点击 图标执行文件,例如下图高亮④所示。
图标执行文件,例如下图高亮④所示。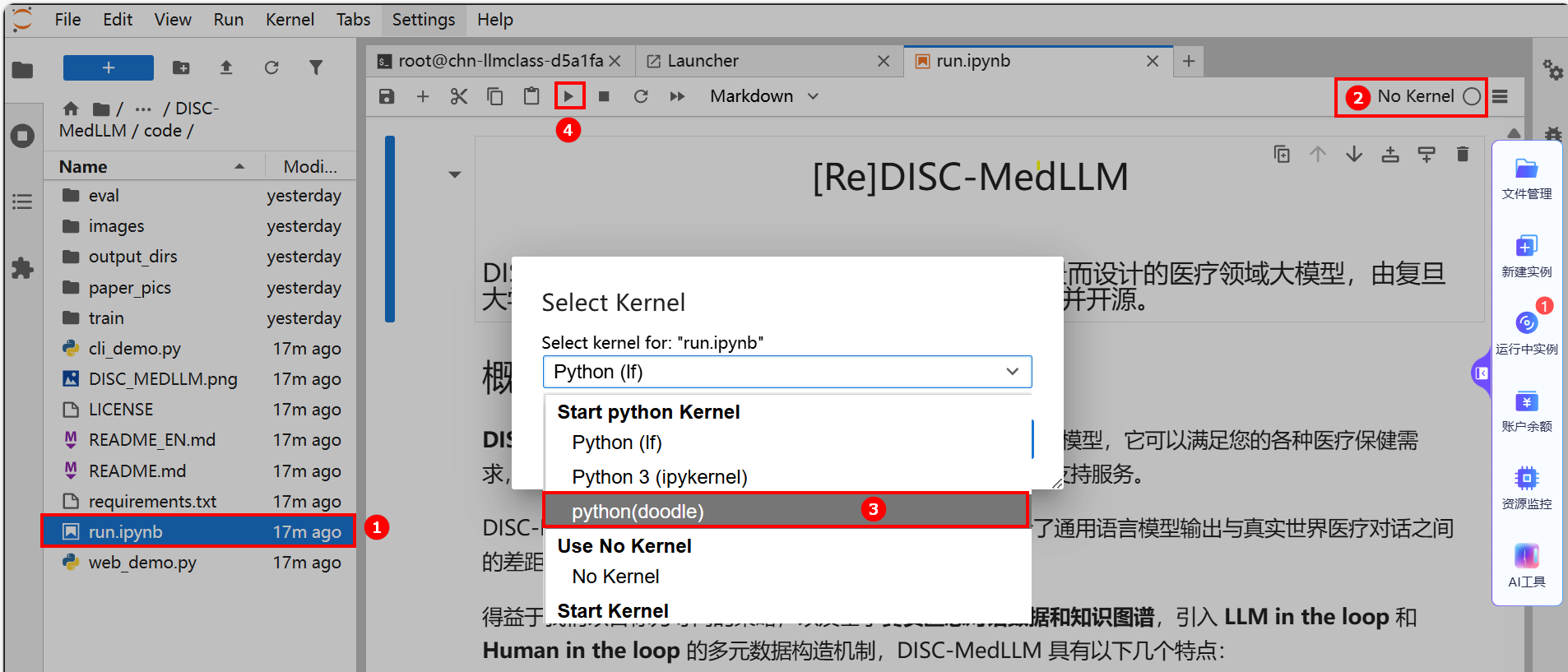
-
如果您使用自定义环境进行论文复现,且为首次操作,请运行以下命令配置环境。
- 运行如下命令创建一个名称为“py39”的Python环境。
conda create -n py39 python=3.9- 运行如下所示的命令激活自定义的环境。
conda activate py39- 运行如下所示的命令,在已经激活的Python环境中安装
ipykernel包。
pip install ipykernel -i https://pypi.tuna.tsinghua.edu.cn/simple- 运行如下所示的命令,将当前Python环境注册为Jupyter内核。
kernel_install --name py39 --display-name "Python 3.9 (py39)"提示您需在对应的Conda环境中运行上述注册命令,否则内核(kernel)将无法正确注册。
-
刷新页面,单击
 图标返回Launcher页面,单击选择Notebook板块下已注册的环境,本例选择上步已激活的 "Python 3.9 (py39)" 内核,例如下图所示。
图标返回Launcher页面,单击选择Notebook板块下已注册的环境,本例选择上步已激活的 "Python 3.9 (py39)" 内核,例如下图所示。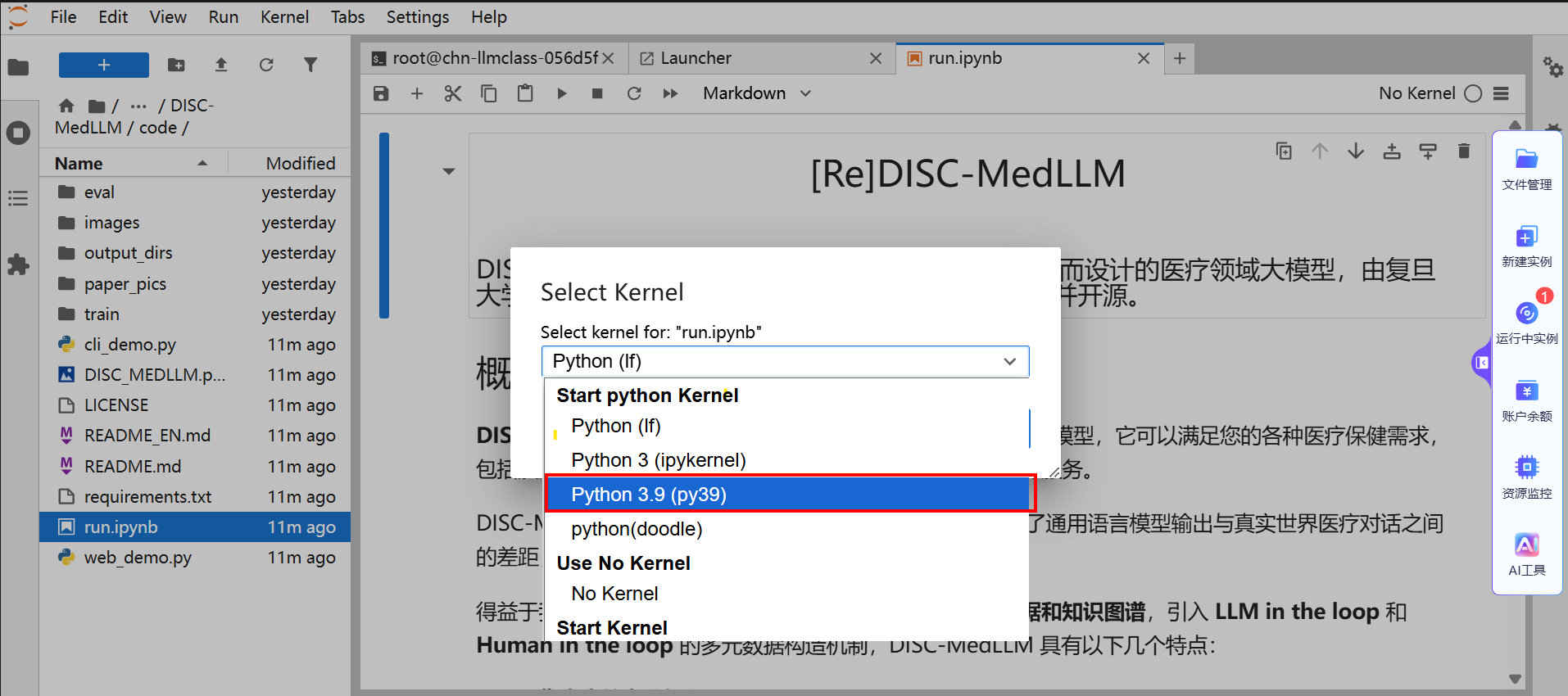
-
切换至
run.ipynb,如下图高亮①所示;点击右上角(如下图高亮②所示),选择“Python 3.9 (py39)”环境,如下图高亮③所示;点击 图标执行文件,例如下图高亮④所示。
图标执行文件,例如下图高亮④所示。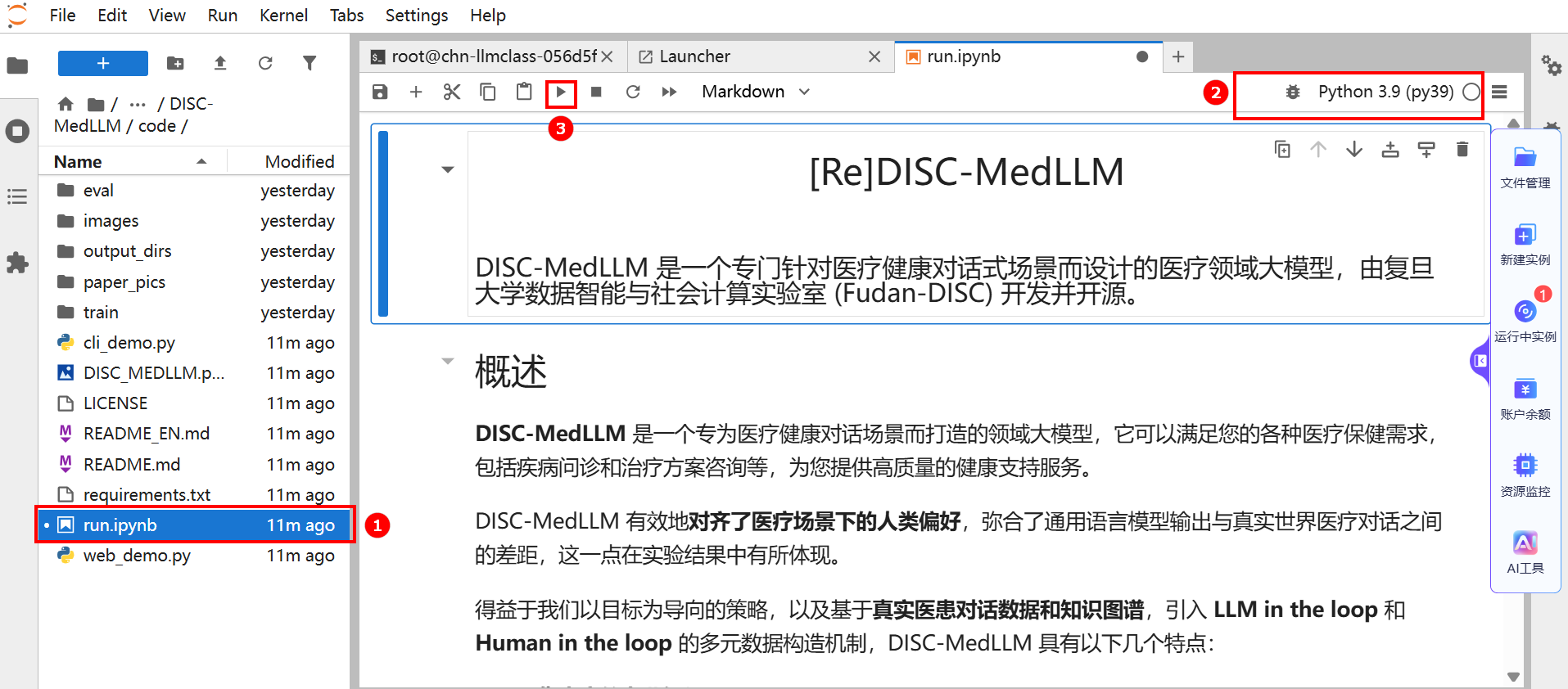
-
脚本运行过程中可打开
codelab/DISC-MedLLM/code/output_dirs目录查看输出文件,该目录下有“DISC_MedLLM”和“logs”两个目录。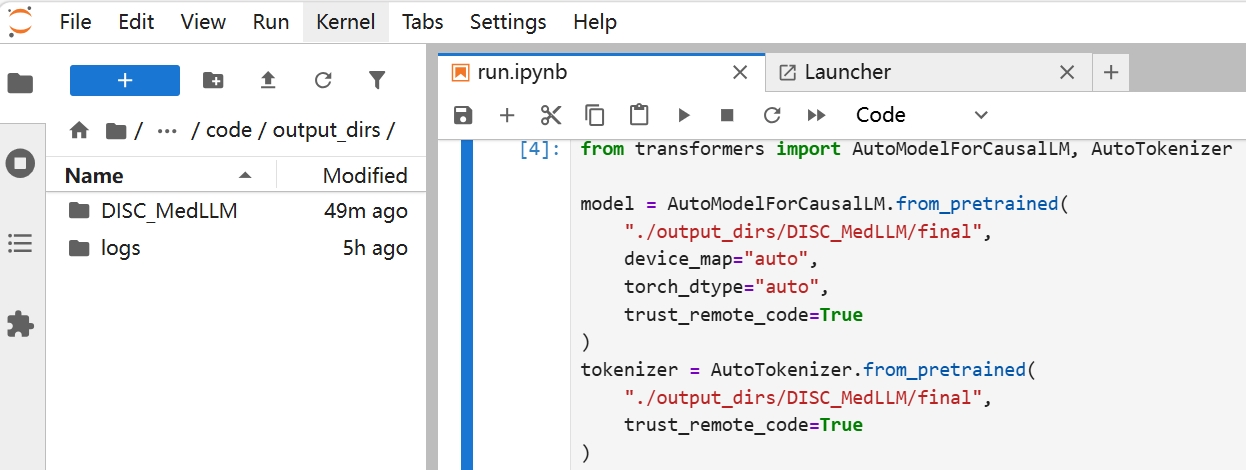
- DISC_MedLLM
- logs
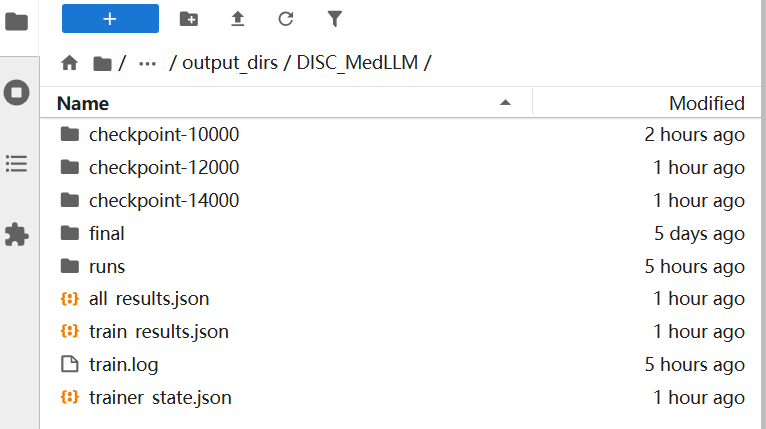
DISC_MedLLM目录存储项目的实验结果,例如下图所示。
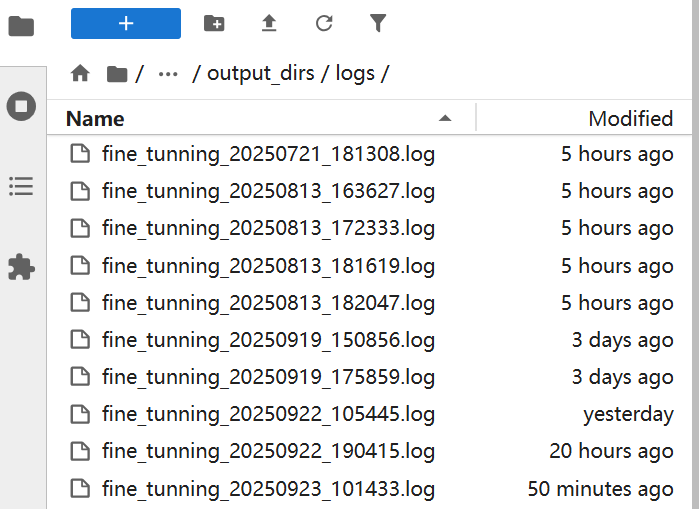
logs目录,存储项目复现过程中的日志。日志文件如下图所示。
查看结果
-
根据日志提示,在模型微调完成后,返回至
codelab/DISC-MedLLM/code/run.ipynb文件,单击运行按钮,如下图高亮①所示,加载已微调完成的模型checkpoint(检查点),如下图高亮②所示。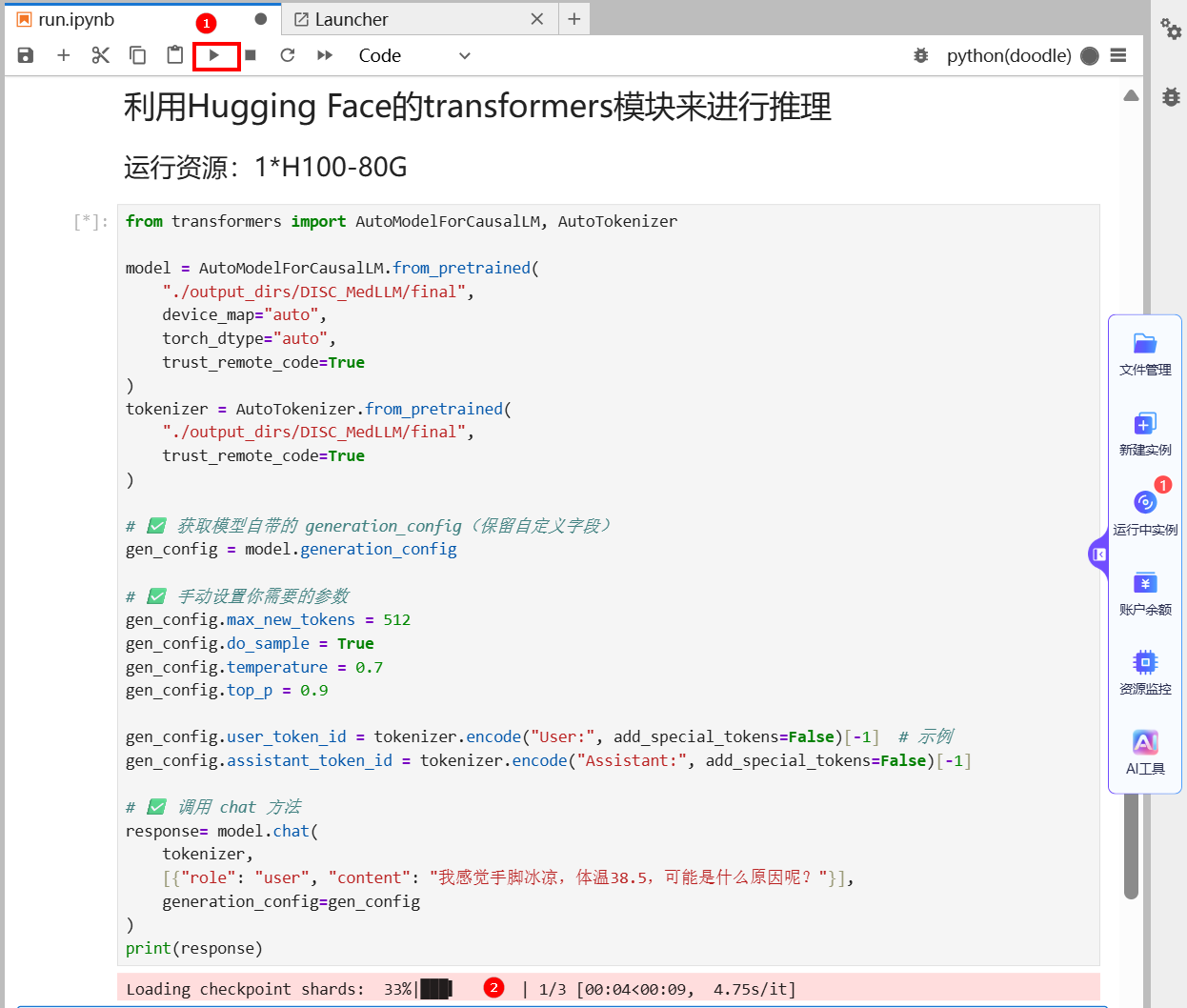
-
检查点加载完成后,系统将基于训练后的模型生成推理结果,如下图所示。

总结
上述流程完整呈现了项目的复现路径。用户可依据本方案的步骤与方法,在项目列表中选择感兴趣的内容,参照对应项目的README文件完成环境搭建、根据.ipynb可运行文件完成模型微调及推理等环节。该方法显著降低了环境配置的复杂度,有助于将精力聚焦于算法理解、实验验证与创新优化,具备较强的实用性与可推广性。